При заданном наборе переменных-предикторов p и переменной отклика множественная линейная регрессия использует метод, известный как метод наименьших квадратов, для минимизации суммы квадратов остатков (RSS):
RSS = Σ(y i – ŷ i ) 2
куда:
- Σ : греческий символ, означающий сумму
- y i : Фактическое значение отклика для i -го наблюдения
- ŷ i : прогнозируемое значение отклика на основе модели множественной линейной регрессии.
Однако, когда переменные-предикторы сильно коррелированы, мультиколлинеарность может стать проблемой. Это может привести к тому, что оценки коэффициентов модели будут ненадежными и будут иметь высокую дисперсию.
Один из способов избежать этой проблемы — вместо этого использовать регрессию основных компонентов , которая находит M линейных комбинаций (известных как «главные компоненты») исходных p предикторов, а затем использует метод наименьших квадратов для подбора модели линейной регрессии, используя главные компоненты в качестве предикторов.
В этом руководстве представлен пошаговый пример выполнения регрессии основных компонентов в R.
Шаг 1: Загрузите необходимые пакеты
Самый простой способ выполнить регрессию основных компонентов в R — использовать функции из пакета pls .
#install pls package (if not already installed)
install.packages(" pls ")
load pls package
library(pls)
Шаг 2: Подгонка модели ПЦР
В этом примере мы будем использовать встроенный набор данных R под названием mtcars , который содержит данные о различных типах автомобилей:
#view first six rows of mtcars dataset
head(mtcars)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3.460 20.22 1 0 3 1
Для этого примера мы подберем модель регрессии основных компонентов (PCR), используя hp в качестве переменной отклика и следующие переменные в качестве переменных-предикторов:
- миль на галлон
- дисп
- дрянь
- вес
- qсек
Следующий код показывает, как подогнать модель PCR к этим данным. Обратите внимание на следующие аргументы:
- scale=TRUE : это говорит R, что каждая из переменных-предикторов должна быть масштабирована, чтобы иметь среднее значение 0 и стандартное отклонение 1. Это гарантирует, что никакая переменная-предиктор не будет чрезмерно влиять на модель, если она измеряется в разных единицах. .
- validation="CV" : это говорит R использовать перекрестную проверку k-fold для оценки производительности модели. Обратите внимание, что по умолчанию используется k=10 кратностей. Также обратите внимание, что вместо этого вы можете указать «LOOCV», чтобы выполнить перекрестную проверку с исключением одного .
#make this example reproducible
set.seed(1)
#fit PCR model
model <- pcr(hp~mpg+disp+drat+wt+qsec, data=mtcars, scale= TRUE , validation=" CV ")
Шаг 3: Выберите количество основных компонентов
После того, как мы подогнали модель, нам нужно определить количество основных компонентов, которые стоит сохранить.
Это можно сделать, взглянув на среднеквадратичную ошибку теста (среднеквадратическую ошибку теста), рассчитанную с помощью k-кратной перекрестной проверки:
#view summary of model fitting
summary(model)
Data: X dimension: 32 5
Y dimension: 32 1
Fit method: svdpc
Number of components considered: 5
VALIDATION: RMSEP
Cross-validated using 10 random segments.
(Intercept) 1 comps 2 comps 3 comps 4 comps 5 comps
CV 69.66 44.56 35.64 35.83 36.23 36.67
adjCV 69.66 44.44 35.27 35.43 35.80 36.20
TRAINING: % variance explained
1 comps 2 comps 3 comps 4 comps 5 comps
X 69.83 89.35 95.88 98.96 100.00
hp 62.38 81.31 81.96 81.98 82.03
В выводе есть две интересующие таблицы:
1. ПРОВЕРКА: RMSEP
Эта таблица сообщает нам среднеквадратичное отклонение теста, рассчитанное с помощью k-кратной перекрестной проверки. Мы можем видеть следующее:
- Если мы используем в модели только термин перехвата, тестовое среднеквадратичное отклонение равно 69,66 .
- Если мы добавим первый основной компонент, тестовый RMSE упадет до 44,56.
- Если мы добавим второй основной компонент, тестовый RMSE упадет до 35,64.
Мы видим, что добавление дополнительных главных компонентов действительно приводит к увеличению RMSE теста. Таким образом, представляется оптимальным использовать в окончательной модели только два основных компонента.
2. ОБУЧЕНИЕ: % объясненной дисперсии
Эта таблица сообщает нам процент дисперсии переменной отклика, объясненной главными компонентами. Мы можем видеть следующее:
- Используя только первый главный компонент, мы можем объяснить 69,83% вариации переменной отклика.
- Добавляя второй главный компонент, мы можем объяснить 89,35% вариации переменной отклика.
Обратите внимание, что мы всегда сможем объяснить большую дисперсию, используя больше основных компонентов, но мы можем видеть, что добавление более двух основных компонентов фактически не увеличивает процент объясненной дисперсии на много.
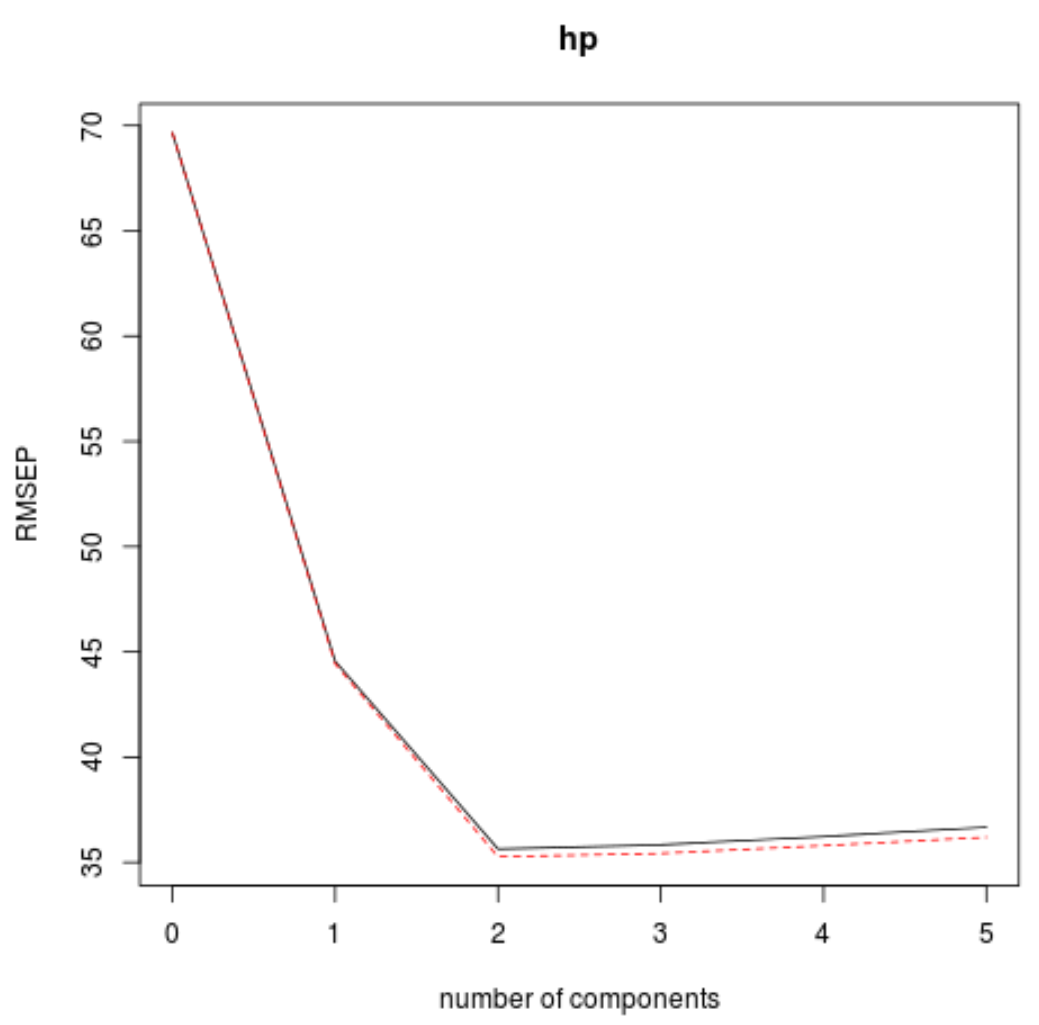
Мы также можем визуализировать среднеквадратичное отклонение теста (вместе с тестовым среднеквадратичным отклонением и R-квадратом) на основе количества основных компонентов с помощью функции validationplot() .
#visualize cross-validation plots
validationplot(model)
validationplot(model, val.type="MSEP")
validationplot(model, val.type="R2")
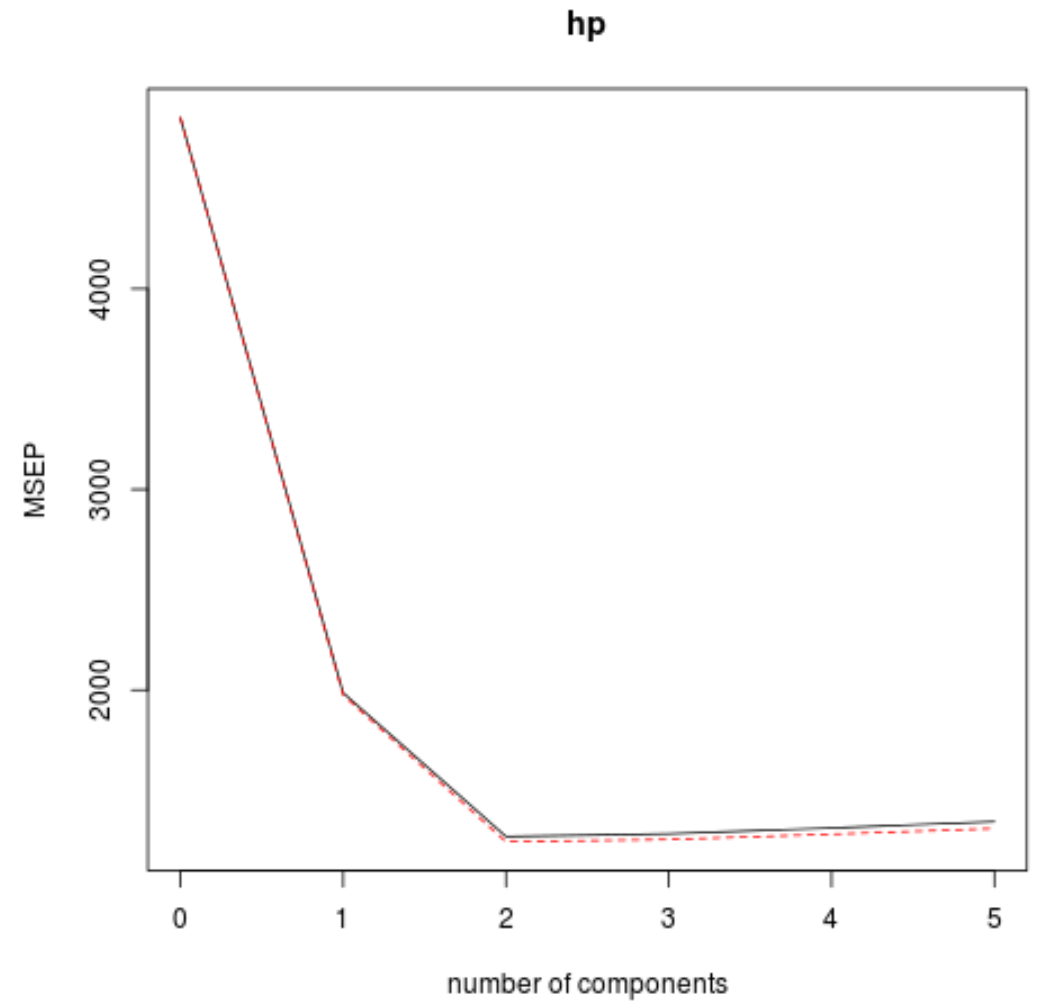
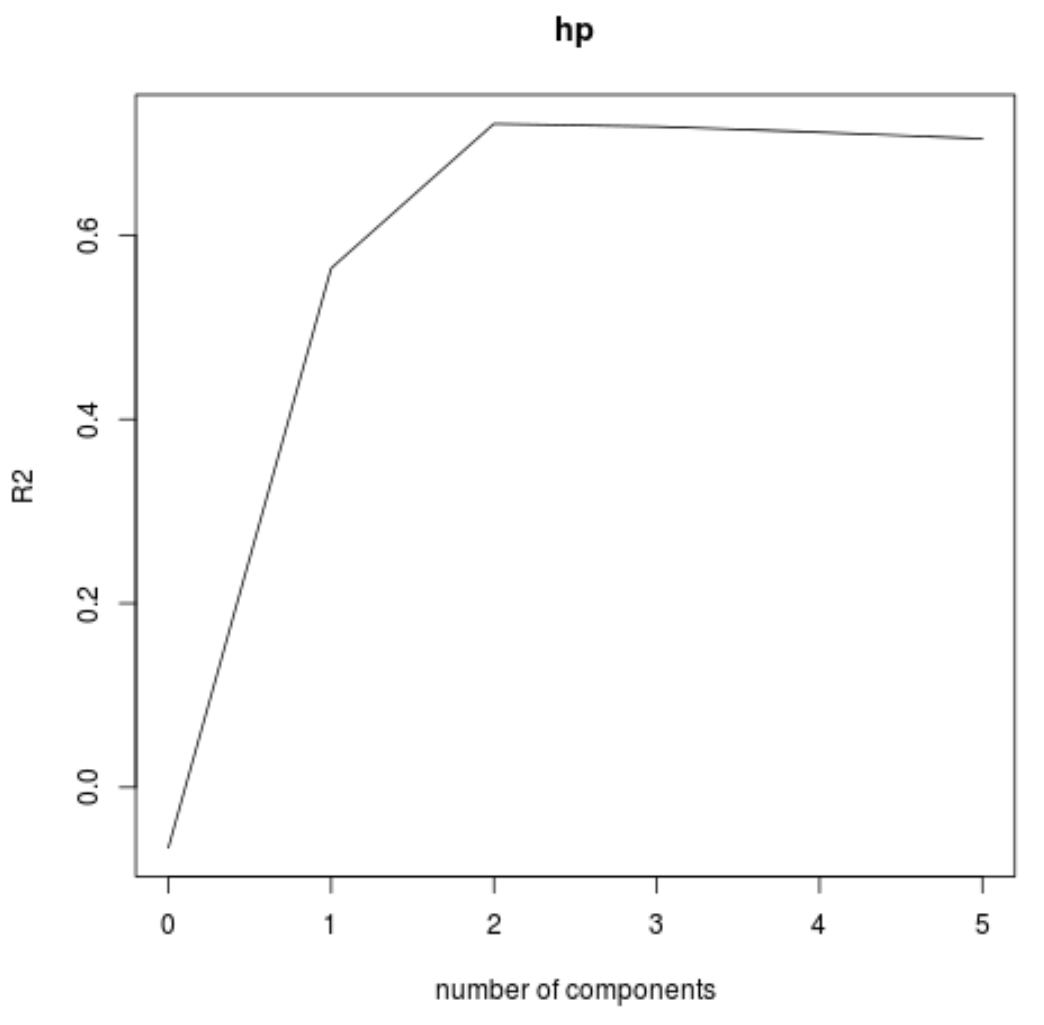
На каждом графике мы видим, что соответствие модели улучшается при добавлении двух основных компонентов, но имеет тенденцию к ухудшению при добавлении большего количества основных компонентов.
Таким образом, оптимальная модель включает только первые две главные компоненты.
Шаг 4: Используйте окончательную модель для прогнозирования
Мы можем использовать окончательную модель PCR с двумя основными компонентами, чтобы делать прогнозы по новым наблюдениям.
В следующем коде показано, как разделить исходный набор данных на набор для обучения и тестирования и использовать модель PCR с двумя основными компонентами для прогнозирования набора для тестирования.
#define training and testing sets
train <- mtcars[1:25, c("hp", "mpg", "disp", "drat", "wt", "qsec")]
y_test <- mtcars[26: nrow (mtcars), c("hp")]
test <- mtcars[26: nrow (mtcars), c("mpg", "disp", "drat", "wt", "qsec")]
#use model to make predictions on a test set
model <- pcr(hp~mpg+disp+drat+wt+qsec, data=train, scale= TRUE , validation=" CV ")
pcr_pred <- predict(model, test, ncomp= 2 )
#calculate RMSE
sqrt ( mean ((pcr_pred - y_test)^2))
[1] 56.86549
Мы видим, что тестовый RMSE оказался равным 56,86549.Это среднее отклонение между прогнозируемым значением hp и наблюдаемым значением hp для наблюдений в тестовом наборе.
Полное использование кода R в этом примере можно найти здесь .